La aleatorización mendeliana es un método epidemiológico propuesto para controlar las asociaciones espurias en los estudios observacionales. Estas asociaciones suelen estar causadas por la confusión derivada de factores sociales, ambientales y de comportamiento, que pueden ser difíciles de medir. La aleatorización mendeliana se basa en la selección de variantes genéticas que se utilizan como variables instrumentales, y que influyen en los patrones de exposición o están asociadas con un fenotipo intermedio de la enfermedad. El presente trabajo pretende discutir cómo seleccionar las variantes genéticas adecuadas como variables instrumentales y presentar las potenciales herramientas metodológicas para lidiar con las limitaciones propias de este método epidemiológico. El uso de variables instrumentales para exposiciones modificables tiene el potencial de atenuar los efectos de limitaciones comunes, como la confusión, cuando se eligen variantes genéticas robustas como variables instrumentales.
The Mendelian randomization is an epidemiologic method proposed to control for spurious associations in observational studies. These associations are commonly caused by confusion derived from social, environmental, and behavioral factors, which can be difficult to measure. Mendelian randomization is based on the selection of genetic variants that are used as instrumental variables that influence exposure patterns or are associated with an intermediate phenotype of the disease. The present work aims to discuss how to select the appropriate genetic variants as instrumental variables and to present methodological tools to deal with the limitations of this epidemiological method. The use of instrumental variables for modifiable exposures has the potential to mitigate the effects of common limitations, such as confusion, when robust genetic variants are chosen as instrumental variables.
Es conocido que un ensayo clínico aleatorizado puede proveer evidencia rigurosa de causalidad debido a su aleatorización y ocultamiento de la asignación; sin embargo, para muchos tipos de exposición es muy costoso, poco práctico o no ético. Cuando un ensayo clínico aleatorizado no es plausible, la aleatorización mendeliana puede ser una alternativa para controlar por confusores en los estudios observacionales1; por ello, el objetivo de esta nota metodológica es discutir la fundamentación, los supuestos, los alcances y las limitaciones relacionadas con este abordaje metodológico.
Fundamentos y supuestos de la aleatorización mendelianaLa aleatorización mendeliana se basa en la segunda y tercera leyes de Mendel (segregación y asignación independiente), las cuales establecen que los dos alelos de cada gen se separan entre sí y de forma independiente durante la gametogénesis, de modo que cada padre solo puede transmitir un alelo, además de que los alelos se distribuyen independientemente en la siguiente generación2.
Las variantes genéticas pueden utilizarse como variables instrumentales (factores que están correlacionados con la exposición, pero son externos a la relación exposición-desenlace) debido a que, al ser asignadas aleatoriamente durante la gametogénesis, aseguran la dirección de causalidad, y al ser heredadas de manera independiente pueden simular la asignación aleatoria realizada en los ensayos clínicos aleatorizados3.
La aleatorización mendeliana puede permite evaluar las asociaciones causales a partir de análisis monogénicos y poligénicos; en los primeros, es posible evaluar la naturaleza causal de los factores de riesgo utilizando variantes de una sola región genética, mientras que en los segundos se incluyen variantes genéticas de múltiples regiones1.
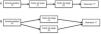
Una vez comprobado qué variante genética puede utilizarse como variable instrumental genética, la población a estudiar se divide en subgrupos según la presencia o la ausencia de la variante genética de riesgo o de protección. Estos subgrupos definidos genéticamente pueden considerarse como análogos a las ramas de un ensayo clínico aleatorizado: los individuos con genotipos de riesgo/protección son análogos a los incluidos en un grupo de intervención, y los individuos sin este genotipo son análogos a los de un grupo de control4,5 (fig. 1).
Bases de datos y análisisLa publicación de bases de datos de libre acceso por parte de los grandes grupos de investigación, como la Genome Aggregation Database (gnomAD)6, y el desarrollo de paquetes específicos de análisis de aleatorización mendeliana (http://www.mrbase.org) han facilitado la implementación de estos estudios.
Los tres supuestos sobre la variable instrumental genética que se utilizará para obtener una estimación no sesgada del efecto causal son: 1) debe ser reproducible y estar fuertemente asociada con la exposición, 2) debe ser independiente de los factores de confusión que sesgan la asociación exposición-desenlace, y 3) solo debe asociarse con el desenlace a través de la exposición estudiada3,4.
Para probar el primer supuesto debe existir evidencia científica previa que haya reportado la asociación en estudio; para el segundo, es necesario analizar las asociaciones entre la variable instrumental genética y los factores de confusión observados cuando estén disponibles; y para el tercero, debe evaluarse la probabilidad de que la variante afecte el desenlace de otra manera que no sea a través de la exposición4.
Amenazas y limitacionesExisten amenazas específicas que pueden conducir a la violación de los tres supuestos básicos4; sin embargo, cada amenaza debe considerarse en el contexto del campo de investigación específico para encontrar el método más adecuado que debe aplicarse.
Variable instrumental genética débilSe considera como una «variable instrumental genética débil»4 si en la regresión de la exposición en la variable genética se identifica un valor del estadístico F<10. Una asociación débil entre la variable instrumental genética y la exposición puede ser engañosa, por lo que la solución propuesta para este escenario es utilizar múltiples variables instrumentales genéticas o diseñar un instrumento de puntuación combinando múltiples variables instrumentales genéticas, como el puntaje alélico o puntaje de riesgo genético (ponderado o no ponderado)3,4.
Estratificación de la poblaciónLa estratificación de la población puede resultar en confusión de la asociación en virtud de diferencias étnicas, pues la frecuencia con que ocurren el alelo y la enfermedad, o las tasas de exposición, pueden variar entre diferentes subgrupos poblacionales4. Por ejemplo, en un estudio de casi 5000 nativos americanos Pima y de la tribus Papago se encontró una fuerte asociación inversa entre el haplotipo HLA Gm Gm3 y la diabetes tipo 2 (odds ratio: 0.27); no obstante, cuando se llevaron a cabo los análisis por estratos étnicos no se encontró asociación7. Por esta razón, es recomendable realizar análisis estratificados en poblaciones heterogéneas, incluso en poblaciones con ascendencia similar4.
Desequilibrio de ligamientoEl desequilibrio de ligamiento ocurre cuando existe una asociación no aleatoria entre diferentes variantes genéticas que están ubicadas cerca en el mismo cromosoma, y que generalmente se heredan juntas. El desequilibrio de ligamiento suele afectar solo a variantes genéticas cercanas y, por lo tanto, rara vez es motivo de preocupación cuando solo se utiliza una variante genética por gen. Una solución puede ser seleccionar únicamente variantes independientes como instrumentos o ajustar las otras variantes genéticas en el análisis de aleatorización mendeliana4.
CanalizaciónEs un proceso mediante el cual la asociación genotipo-enfermedad esperada es amortiguada por variantes genéticas adaptativas. Este proceso es difícil de estudiar en la práctica por el hecho de que el organismo siempre trata de mantener la homeostasis. Se podría asumir que existen muchos genes y vías metabólicas que, a su vez, podrían estar activados en diferente magnitud, con el fin de contrarrestar los efectos negativos de la variable instrumental genética empleada en la aleatorización mendeliana4.
PleiotropíaEn la práctica, muchas variantes genéticas son pleiotrópicas, es decir, se asocian con múltiples factores de riesgo4. Los posibles mecanismos por los cuales una variante genética puede estar asociada con múltiples factores de riesgo se dividen en pleiotropía vertical y pleiotropía funcional (fig. 2). Estos dos casos no se excluyen mutuamente, pues es posible que los dos estén presentes en la misma variante8. Con el fin de interpretar las estimaciones del efecto de causalidad, se asume el efecto de cada uno de los factores de riesgo como «causalmente independientes»8.
Pleiotropía vertical y pleiotropía funcional. A: En la pleiotropía vertical, la variante genética “A” se asocia con un factor de riesgo (1), la variante genética “A” junto con el factor de riesgo (1) se asocian a otro factor de riesgo (2), y en conjunto llevan al desenlace “Y”. B: En la pleiotropía funcional, la variante genética “A” se asocia con dos factores de riesgo (1 y 2) simultáneamente, y en conjunto la variante genética “A”, el factor de riesgo (1) y el factor de riesgo (2) llevan al desenlace “Y”.
En la actualidad existen métodos de análisis robustos para detectar y ajustar la pleiotropía cuando se utilizan múltiples variables instrumentales genéticas9. El análisis de dos muestras se ha vuelto atractivo debido a que aumenta el tamaño muestral, gana poder estadístico, estima asociaciones causales basadas en diferentes variantes genéticas y evalúa los supuestos básicos de la aleatorización mendeliana10. Los métodos más utilizados son el MR-Egger, para el control de sesgos introducidos por las variantes genéticas, y el MR-PRESSO, que elimina las múltiples variantes genéticas del análisis cuya estimación causal específica de la variante difiere sustancialmente de las de otras variantes9.
ConclusionesLa aleatorización mendeliana es un método de análisis que permite probar hipótesis de asociaciones causales en estudios observacionales mediante el uso de variantes genéticas como variables instrumentales; no obstante, para que el abordaje sea válido, las variables instrumentales genéticas deben cumplir con los supuestos básicos, además de considerar las posibles amenazas a la violación de estos.
Esta nota metodológica pretende permitir al lector identificar que la aleatorización mendeliana es una alternativa metodológica viable que facilita lidiar con la confusión cuando se eligen variantes genéticas robustas como variables instrumentales.
Editor responsable del artículoCarlos Álvarez Dardet.
Contribuciones de autoríaTodas las personas firmantes participaron en la elaboración de la nota metodológica. M. Ancira y H. Lamadrid-Figueroa escribieron la primera versión, que fue revisada y editada por N. Smith. Todas las personas firmantes han leído y aprobado la versión final antes de ser enviada para su publicación.
FinanciaciónNinguna.
Conflictos de interesesNinguno.