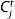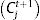Estimar los casos diarios de COVID-19 durante el primer año de pandemia en Santiago Región Metropolitana (SRM) y en Chile con cifras más realistas que las registradas oficialmente.
MétodoEstimación retrospectiva de los casos diarios a partir de los datos diarios de fallecimientos por COVID-19, un estudio de seroprevalencia y el algoritmo REMEDID (Retrospective Methodology to Estimate Daily Infections from Deaths).
ResultadosEn SRM se observa que: 1) el pico máximo de contagios fue superior al doble del registrado en las estadísticas oficiales; 2) dicho pico se alcanzó el 22 de mayo de 2020 (IC95%: 20 a 24 de mayo), esto es, 24 días antes de la fecha oficial del pico de contagios; y 3) el primer contagio estimado tuvo lugar el 28 de enero de 2020 (IC95%: 21 de enero a 16 de febrero), esto es, 36 días antes de la fecha oficial. En Chile, la situación es similar. Durante la primera ola, SRM concentró el 70-76% de los infectados de Chile, mientras que a partir de agosto de 2020 solo concentró el 36-39%.
ConclusionesLos registros oficiales de casos de COVID-19 en SRM y Chile infravaloraron el número real de positivos y mostraron un retraso de alrededor de un mes en la dinámica de contagios. Esta no es una situación aislada, ya que se sabe que también ha sido así en otros países. No obstante, es importante disponer de estimaciones fidedignas para una correcta modelización de la expansión del virus.
Estimate daily infections of COVID-19 during the first year of the pandemic in the Santiago Metropolitan Region (SRM) in Chile and Chile that are more realistic than those officially registered.
MethodRetrospective estimate of daily infections from daily data on COVID-19 deaths, a seroprevalence study, and the REMEDID (Retrospective Methodology to Estimate Daily Infections from Deaths) algorithm.
ResultsIn SRM, it is observed that: 1) the maximum peak of infections was more than double that registered in the official statistics; 2) such peak was reached on May 22 (95% CI: 20–24 May), 2022, that is, 24 days before the official date of the peak of infections; and 3) the first estimated contagion took place on January 28, 2020 (95% CI: January 21 to February 16), that is, 36 days before the official date. In Chile, the situation is similar. During the first wave SRM accounted for 70%–76% of those infected in Chile, while from August 2020 onwards it accounted for 36%–39%.
ConclusionsThe official records of COVID-19 infections in SRM and Chile underestimated the real number of positives and showed a delay of about a month in the dynamics of infections. This is not an isolated situation, as it is known to have been the case in other countries as well. However, it is important to have reliable estimates for a correct modeling of the spread of the virus.
A finales de 2019 apareció el SARS-CoV-2 y a principios de 2020 provocó una pandemia que, 3 años después, en abril de 2023, ya ha causado más de 764 millones de casos en todo el mundo y 6,9 millones de muertes1. La modelización matemática de la COVID-19ha permitido anticipar la evolución de la pandemia, facilitando la adaptación. Por ejemplo, en el Reino Unido, el gobierno era reticente a imponer restricciones. Sin embargo, el 16 de marzo de 2020, el Imperial College of London publicó un informe en el que se estimaba que habría medio millón de muertos en el Reino Unido si no se aplicaban medidas de mitigación2. Ese mismo día, el gobierno recomendó trabajar desde casa y evitar viajes innecesarios, y una semana después decretó el confinamiento nacional3. La evolución de la pandemia se monitoriza mediante el registro de contagios y de muertes asociadas, lo cual resultó extremadamente difícil al principio de la pandemia, ya que un tercio de los infectados no presentaban síntomas4 y hubo escasez generalizada de pruebas de diagnóstico. Por lo tanto, los estudios retrospectivos son importantes para entender la dispersión original del virus. Barber et al.5 modelizaron estadísticamente las infecciones diarias en 190 países y territorios, y dedujeron que hasta el 14 de noviembre de 2021 hubo cerca de 3390 millones de infecciones, el 44% de la población mundial. Oficialmente solo se habían confirmado 254 millones de infecciones globales6, lo que implica una detección de solo el 7,5% de los casos. También Karlinsky y Kobak7 estudiaron el exceso de mortalidad por cualquier causa respecto a años anteriores en 103 países, y dedujeron 5,2 millones de muertes por COVID-19 hasta julio de 2021, un millón más que las registradas oficialmente. Y localmente la detección pudo ser incluso peor. Sin embargo, en algunos lugares el porcentaje de detección fue mucho más bajo. Por ejemplo, en Damasco, Siria, solo se detectaron un 1,25% (1-3%) de las muertes por COVID-19 en julio y agosto de 20208. En Lusaka, Zambia, se descubrió, mediante la realización sistemática de test en cadáveres, que las infecciones del verano de 2020 fueron subestimadas9. En Mogadiscio, Somalia, se estimó el exceso de muertes a partir de imágenes satelitales, demostrando que las infecciones también habían sido subestimadas y que el virus pudo haber entrado en el país en diciembre de 201910, mucho antes de que se detectara el primer caso en Somalia el 16 de marzo de 202011.
En España se han estimado a posteriori las infecciones diarias de la primera ola mediante el algoritmo REMEDID (Retrospective Methodology to Estimate Daily Infections from Deaths), a partir de las defunciones diarias por COVID-19 y de dos estudios de seroprevalencia12. Este estudio reveló que, cuando se decretó el confinamiento nacional el 14 de marzo de 2020, oficialmente se habían registrado 1832 infecciones ese mismo día, aunque en realidad hubo entre 64.000 y 78.000 infecciones, es decir, de 35 a 42 veces más. En los Estados Unidos de América, la aplicación del mismo algoritmo sugiere que el virus entró en el país el 28 de diciembre de 2019, 16 días antes del primer caso registrado13.
En Chile, el 3 de marzo de 2020 se confirmó el primer caso de COVID-1914 y el 18 de marzo se decretó la cuarentena nacional15. Hasta el 12 de febrero de 2023 se contabilizaron 5.138.732 personas contagiadas y 63.998 fallecidos totales entre casos confirmados y sospechosos16.
En este estudio estimamos las infecciones diarias durante el primer año de pandemia mediante el algoritmo REMEDID en Santiago Región Metropolitana (SRM) y Chile, gracias a los resultados del estudio de seroprevalencia de la SRM17. Los resultados aquí presentados aportan información relevante sobre la propagación del virus, lo que podría ayudar a diseñar planes de actuación para futuras pandemias.
MétodoCon el algoritmo REMEDID12 se pueden estimar los contagios diarios a partir de: 1) la distribución de probabilidad teórica que establece el tiempo transcurrido desde la infección hasta la muerte; 2) el registro diario de los decesos producidos por COVID-19; y 3) la cantidad total de infectados en un tiempo específico, lo cual se calcula con estudios de seroprevalencia. Este método tiene sentido aplicarlo cuando el registro de las muertes es más fiable que el de las infecciones, como es el caso de SRM.
La idea del algoritmo es sencilla: dada una muerte en un determinado día, descontamos los días que pasaron desde la infección hasta la muerte (DIM) para saber la fecha de la infección. Sin embargo, la ejecución del algoritmo se complica, ya que los DIM difieren entre fallecidos, lo que nos obliga a utilizar una variable aleatoria, XDIM, que represente esos DIM. Los DIM de cualquier persona son la suma del periodo de incubación (PI) y del periodo desde la aparición de síntomas hasta la muerte (PSM), es decir, DIM=PI+PSM. Los PI y los PSM también son variables aleatorias, cuyas distribuciones de probabilidad fueron estimadas por Linton et al.18 a partir de registros de los primeros meses de la pandemia en China. El PI fue aproximado con una distribución log normal de 5,6 días (intervalo de confianza del 95% [IC95%]: 5,0-6,3) de media y 5 días (IC95%: 4,4-5,6) de mediana, cuya función de densidad de probabilidad (FDP) llamaremos g(t), y el PSM se aproximó con otra distribución log normal de 14,5 días (IC95%: 12,5-17,0) de media y 13,2 días (IC95%: 11,3-15,3) de mediana, cuya FDP llamaremos h(t). La convolución de g(t) y h(t) se corresponde con la FDP de XDIM, f(t), que aunque no es una distribución log normal se acerca mucho a una log normal con media de 20,1 días y mediana de 18,8 días12. Como los datos con los que vamos a trabajar están en días, usaremos una aproximación de f(t) discretizada en días, F(n), siendo n los días transcurridos desde la infección.
Dada una serie temporal de muertes, x(n), se puede calcular la serie temporal de infecciones diarias, y(n), que ocasionaron dichas muertes como:
En la práctica no podemos calcular el sumatorio infinito. Cuando sea posible truncaremos en 60 días, ya que el 99,99% de XDIM será menor que dicho valor. Al final de la serie eliminaremos un mínimo de 33 días, que es la cota superior del 95% de los valores de XDIM. Por lo tanto, la serie temporal de infecciones acabará 33 días antes que la serie de muertes. Para obtener la serie de infecciones totales, y no solo las que produjeron las muertes, debemos conocer la tasa de letalidad (CFR, case fatality rate) y aplicar la fórmula:
Estimación de erroresLos parámetros que definen las FDP de PI y PSM presentan una incertidumbre en forma de intervalos de confianza, aunque no se conoce la distribución de los errores dentro de dichos intervalos. Aquí asumiremos que siguen una distribución uniforme, es decir, que todos los valores de los intervalos de confianza son igualmente probables. Para estimar la propagación de los errores realizaremos una simulación de Monte Carlo con 10.000 posibles combinaciones aleatorias de los parámetros que definen las FDP de PI y PSM. El algoritmo REMEDID se aplicará con dichas FDP y los extremos de los IC95% se obtendrán con los cuartiles 2,5 y 97,5 de los resultados de las 10.000 simulaciones. En general, los resultados se presentarán con el valor promedio de las simulaciones acompañado del IC95%.
DatosExisten dos registros de muertes diarias para el periodo 2020-2021:
- •
Departamento de Estadísticas e Información de Salud (DEIS)19, que forma parte de la División de Planificación Sanitaria y tiene datos de muertes por COVID-19 para SRM y todo Chile durante el periodo 2020-2023.
- •
Ministerio de Ciencia (MC, Min Ciencia/Datos COVID-1920), con datos de muertes por COVID-19 para Chile. Los datos para SRM y el resto de regiones se pueden descargar desde el Ministerio de Ciencias, Tecnología, Conocimiento e Innovación21. Otro producto recoge los datos globales de contagio para todo Chile (Ministerio de Salud)22.
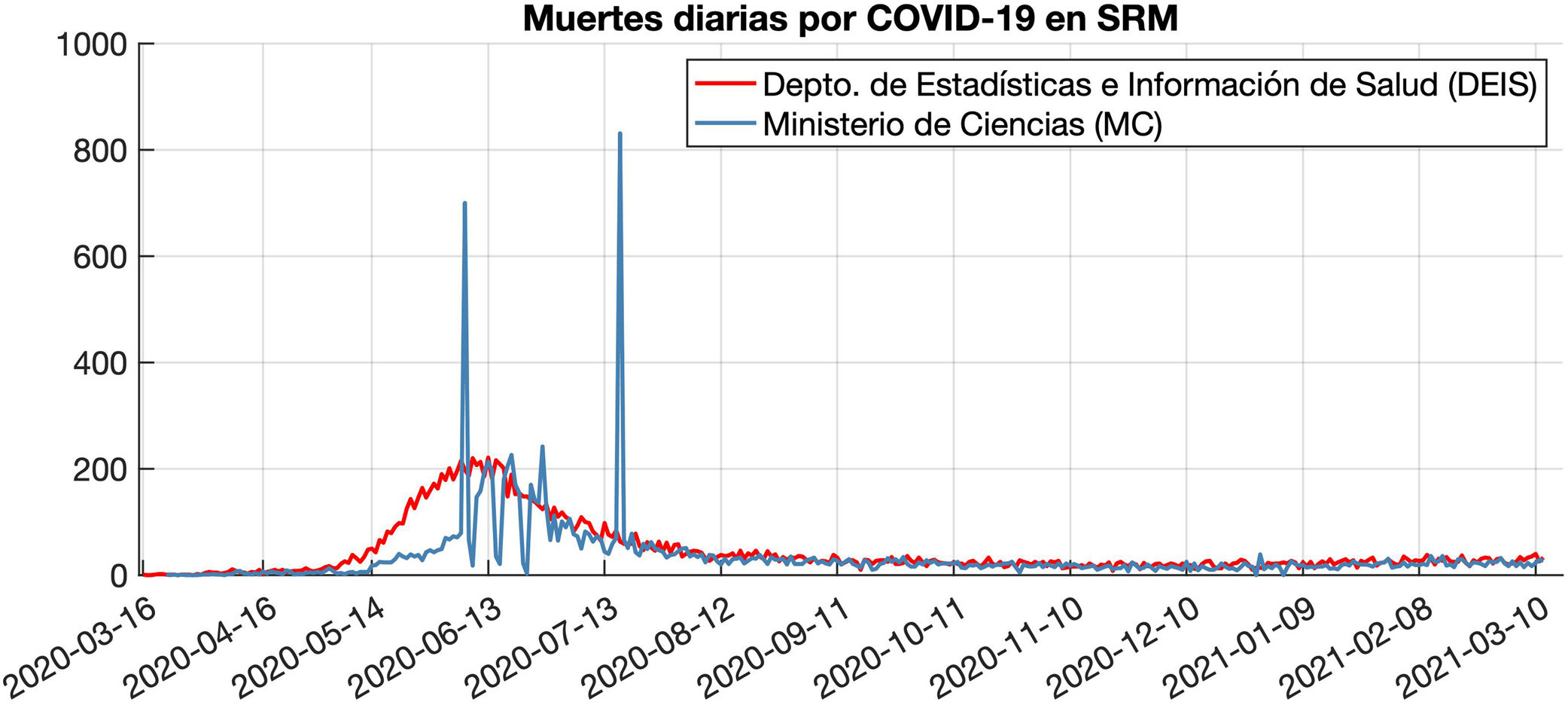
Estos datos fueron descargados entre el 21 y el 28 de junio de 2022. Las dos bases presentan diferencias. Por un lado, el DEIS recoge 15.787 muertes acumuladas hasta el 12 de marzo de 2021, mientras que el MC recoge 12.256. Por otro lado, las gráficas son muy parecidas a partir de julio de 2020, pero hasta esa fecha parece que los datos del MC presentan ciertas anomalías (Fig. 1). Por ejemplo, el MC registró 700 muertes el 7 de junio y 831 el 17 de julio de 2020, y esto no es compatible con las muertes producidas durante las fechas cercanas. Esto podría deberse al registro en dichos días de algunas muertes no registradas en los días o semanas previas, lo cual explicaría el menor número de muertes del MC respecto al DEIS durante el mes de mayo. Además, la primera muerte en el MC se registró 18 días después que la primera en el DEIS. Finalmente, los datos del MC muestran una caída del registro de muertes los fines de semana de junio, lo cual podría deberse al descanso del personal. Este tipo de problemas no se observan en los datos del DEIS, por lo que solo presentaremos los resultados con estos últimos en la aplicación del algoritmo REMEDID.
El cálculo del CFR se realiza mediante la ratio entre las muertes y las infecciones acumuladas hasta el 12 de marzo de 2021, que es cuando acabó el estudio de seroprevalencia en SRM. Entre el 18 de diciembre de 2020 y el 12 de marzo de 2021 se realizaron 1243 test en SRM, revelando 180 casos positivos, es decir, un 14,5%23. Asumiendo este porcentaje de infectados para los 7.213.842 habitantes de SRM24 se obtiene un total de 1.044.643 infectados acumulados hasta el 12 de marzo de 2021. Como hasta esa fecha hubo 15.787 muertes acumuladas según el DEIS, se obtiene un CFR medio del 1,51% para el primer año de pandemia.
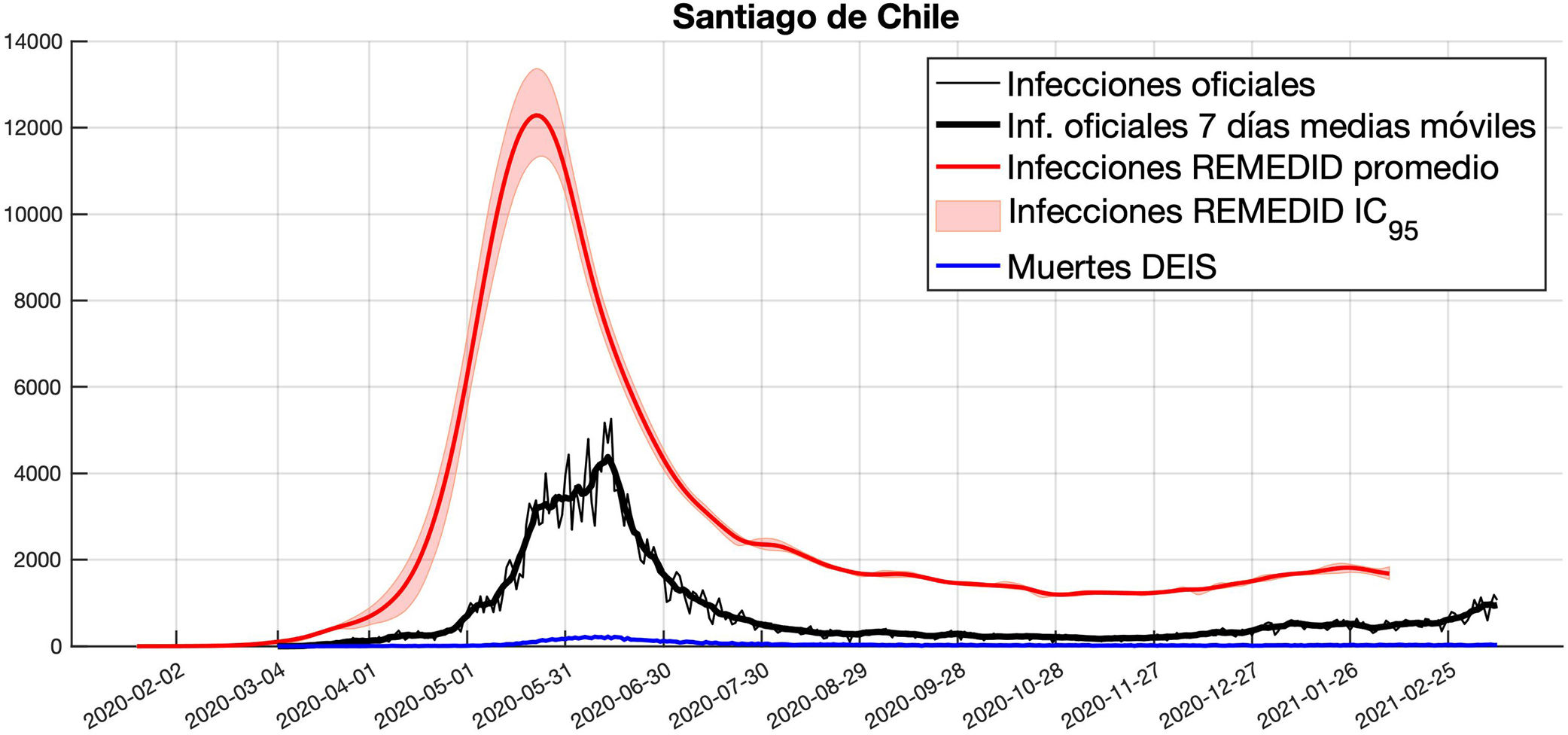
ResultadosSantiago Región MetropolitanaComo resultado del algoritmo REMEDID, a partir de los datos de muertes por COVID-19 en SRM registrados por el DEIS, y usando un CFR del 1,51%, se obtiene una serie temporal (para cada FDP de DIM) de infecciones diarias estimadas, a la que llamaremos «infecciones REMEDID». Para discutir los resultados calculamos el promedio diario de las 10.000 simulaciones de infecciones REMEDID junto con su IC95% asociado (Fig. 2). La serie temporal promedio es prácticamente idéntica a la mediana de las simulaciones. Esta serie temporal acabará 33 días antes del fin del estudio de seroprevalencia, es decir, el 7 de febrero de 2021. Recordemos que las infecciones REMEDID son coherentes con la información teórica de la enfermedad (distribución de probabilidad), los registros de las muertes y el estudio de seroprevalencia, coherencia que no presentan los datos oficiales de infectados. Por ejemplo, los datos oficiales no presentan desfase entre el pico de infecciones y de muertes diarias, cuando teóricamente debería haber un desfase de unos 20 días (Fig. 2). Comparando las infecciones REMEDID y las infecciones oficiales, observamos ciertas diferencias (Fig. 2). Por un lado, la primera infección REMEDID aparece el 28 de enero de 2020 (IC95%: 21 enero a 16 febrero), 36 días (IC95%: 17-43 días) antes de la primera infección registrada oficialmente el 4 de marzo de 2020. Por otro lado, el pico de infecciones REMEDID durante la primera ola es de 12.287 (IC95%: 11.306-13.369) y se alcanza el 22 de mayo de 2020 (IC95%: 20 a 24 de mayo), mientras que el pico de los datos oficiales se alcanza 24 días después (IC95%: 22-26), el 14 de junio, con solo 5264 infectados. Además, los datos oficiales oscilan cada 7 días presentando un mínimo los sábados, igual que pasaba en los datos oficiales de España12. Este ciclo no se debe a ninguna razón epidemiológica y se elimina con unas medias móviles de 7 días. En ese caso, el máximo de la primera ola alcanza los 4378 infectados el 13 de junio, incrementando la diferencia con los infectados REMEDID. Finalmente, las infecciones REMEDID acumuladas hasta el 7 de febrero de 2021 son 1.009.453 (IC95%: 982.098-1.020.542), mientras que las oficiales son 257.181, por lo que solo se detectó un 25,5% (IC95%: 25,2-26,2) de las infecciones durante el primer año de pandemia.
ChileLa dificultad para aplicar REMEDID en el ámbito nacional reside en el desconocimiento del CFR, ya que no se dispone de ningún estudio de seroprevalencia nacional. Para solventar esta dificultad asumiremos que el CFR en el país fue similar al de SRM. Aunque esta suposición parece razonable, no se puede comprobar su veracidad, por lo que los resultados obtenidos estarán supeditados a la veracidad de la hipótesis.
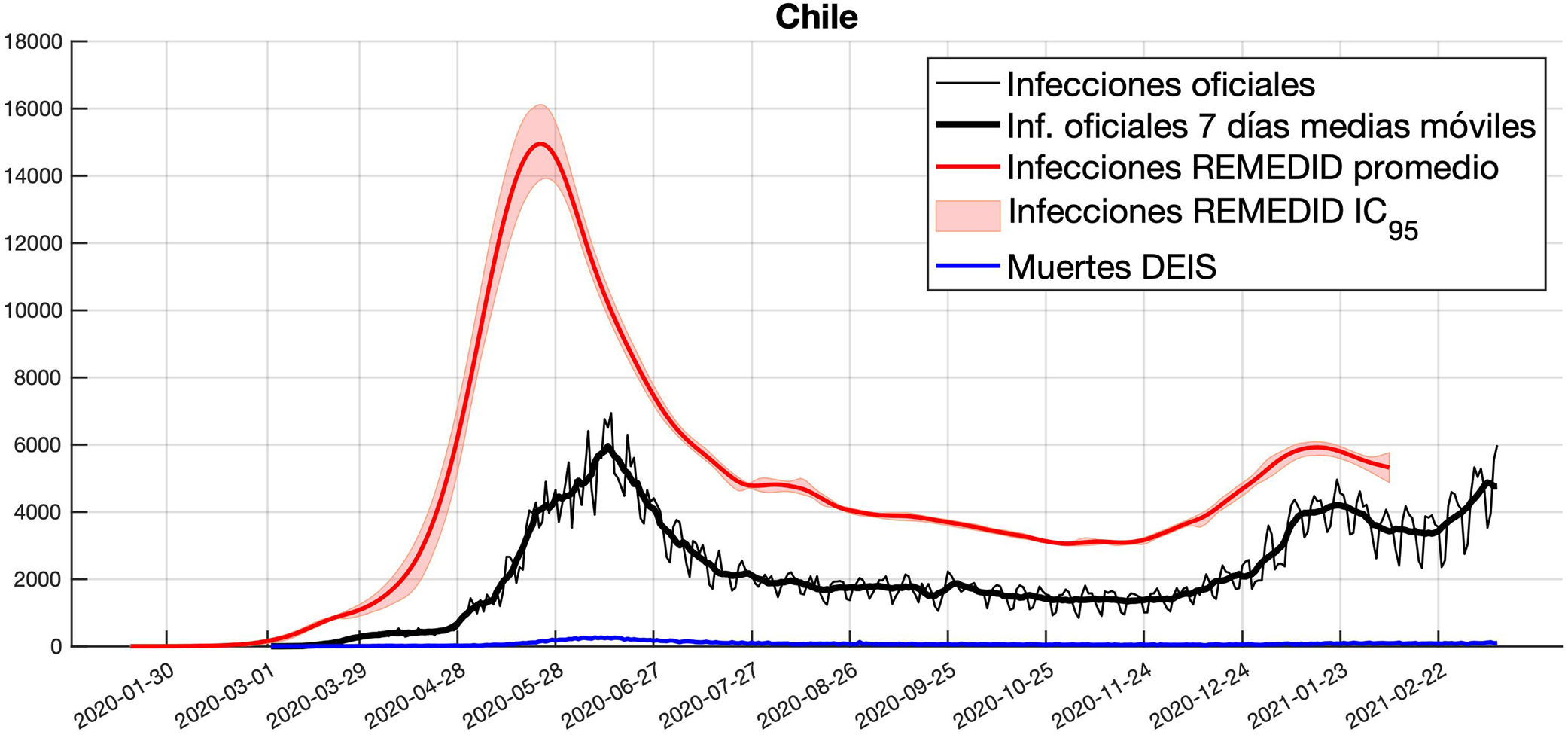
En la figura 3 se muestra la serie promedio de las 10.000 simulaciones de infectados REMEDID, y su IC95%, estimada para Chile considerando un CFR de 1,51%, las muertes diarias del DEIS y las infecciones oficiales. Comparando ambas series de infecciones observamos discrepancias semejantes a las observadas en SRM: 1) la fecha de la primera infección promedio es el 25 de enero de 2020 (IC95%: 21 de enero a 15 de febrero) según REMEDID, 37 días (IC95%: 16-41) antes de la primera infección registrada oficialmente (que data del 2 de marzo); 2) el pico de infecciones REMEDID durante la primera ola es de 14.950 (IC95%: 13.853-16.116) y se alcanza el 23 de mayo de 2020 (IC95%: 21 a 26 mayo), mientras que el pico oficial se alcanza 22 días después (IC95%: 19-24), el 14 de junio, con solo 6938 infectados; 3) los datos oficiales oscilan cada 7 días, y al calcular las medias móviles de 7 días el máximo se alcanza el 13 de junio, con 5.956 infectados. En el ámbito nacional se contabilizaron 720.047 infecciones oficiales hasta el 7 de febrero de 2021, frente a 1.770.720 (IC95%: 1.720.807-1.794.150) infecciones REMEDID. Por lo tanto, solo se detectaron el 40,2% (IC95%: 39,7-41,4) de las infecciones. Este resultado confirma el subregistro de casos estimado a partir de la letalidad ajustada por retraso25.
DiscusiónEste es el primer estudio retrospectivo de infecciones diarias para SRM y Chile, y mejora el conocimiento actual reflejando que: 1) el virus entró en circulación alrededor de un mes antes de lo que se suponía; 2) afectó a más personas de las que se pensaba; y 3) las medidas de mitigación tuvieron efecto antes de lo que reflejaron los registros oficiales. Una situación similar se dio en España, donde la primera infección oficial se registró el 20 de febrero de 2020, un mes y medio después de la primera infección REMEDID, que se fechó el 8 o 9 de enero12. Tras revisar casos antiguos a principios de 2021, las autoridades españolas fecharon el primer caso el 1 de enero de 2020, confirmando la fiabilidad del algoritmo REMEDID. En los Estados Unidos, el primer y el segundo casos se registraron el 13 y el 15 de enero de 202126,27. Sin embargo, al aplicar el algoritmo REMEDID se fijó la primera infección el 28 de diciembre de 201913, 3 días antes de que las autoridades sanitarias de Wuhan informaran de unos casos de neumonía de origen desconocido28. Normalmente los primeros casos son difíciles de detectar porque nadie los busca. Esto también se observó en Chile con la epidemia de gripe AH1N1 de 200929.
Otro problema extendido fue el inframuestreo de las infecciones debido a la escasez de pruebas de detección. En SRM y Chile, el pico de infecciones REMEDID dobló ampliamente al oficial y se alcanzó entre 22 y 24 días antes de lo registrado. El desfase se debe al periodo de incubación y empeoramiento de síntomas antes de la realización de un test, reservados para casos graves. Consecuentemente, el efecto de las medidas de contención se observó con retraso. El 18 de marzo de 2020, el gobierno de Chile declaró el estado constitucional de catástrofe para todo el territorio nacional30, lo que desaceleró las infecciones REMEDID, pero no las oficiales. El 13 de mayo se decretó la cuarentena total en SRM31. El pico de infecciones REMEDID se alcanzó 9 días después, y el oficial 32 días después, dando la sensación de ineficacia del confinamiento.
Las infecciones acumuladas durante la primera ola (hasta el 2 de agosto) fueron 717.712 (IC95%: 698.035-727.083) infecciones REMEDID y 198.729 infecciones oficiales en SRM, y 987.482 (IC95%: 960.524-1.002.756) infecciones REMEDID y 328.176 infecciones oficiales en Chile. Por lo tanto, las infecciones en SRM, con 7,2 millones de habitantes (el 37% de la población de Chile32), representaron el 70-76% del total de las infecciones en el país. La mayor densidad poblacional en SRM respecto al resto del país pudo favorecer la propagación inicial del virus. Esta situación cambia entre el 2 de agosto y el 7 de febrero de 2021, periodo en que SRM acumula 291.741 (IC95%: 283.858-295.657) infecciones REMEDID y 58.986 infecciones oficiales, y Chile acumula 783.238 (IC95%: 760.427-792.370) infecciones REMEDID y 393.952 infecciones oficiales. Es decir, después de la primera ola, las infecciones en SRM bajaron al 36-39% de las infecciones del país. Esto podría deberse a que en esa época las restricciones en SRM fueron más severas que en el resto del país. Por ejemplo, en julio de 2020, la mitad norte de la comuna de Santiago cumplía 100 días de aislamiento ininterrumpidos, una de las cuarentenas más largas del mundo33.
Según las infecciones REMEDID, a finales de noviembre de 2020 arranca la segunda ola en Chile, la cual se extenderá hasta junio de 202134. Posiblemente esta ola se agudizó por el permiso de vacaciones que concedió el gobierno a algunas comunas a partir del 30 de diciembre de 202030, y por el inicio de las vacaciones escolares de verano en todo el país. No obstante, a partir de diciembre de 2020 sería necesario recalcular el CFR para aplicar REMEDID, ya que el 16 de diciembre se aprobó el uso de las vacunas de Pfizer y BioNTech30. Aunque la vacuna no evitaba los contagios, sí disminuía la gravedad de los síntomas y por tanto el CFR. A partir de enero de 2022 sería necesario realizar otra estimación del CFR, ya que se observó un descenso del CFR previo en Chile debido a la presencia de la variante ómicron (B.1.1.529) del virus35. La variante delta (B.1.617.2) también queda fuera del periodo de estudio, ya que fue predominante en Chile en otoño de 202135.
Disponibilidad de bases de datos y material para réplicaLos datos y el software utilizados para el estudio están disponibles en el repositorio de GitHub: https://github.com/isavig/REMEDID.
Al inicio de la pandemia de COVID-19 el registro de infecciones fue muy complicado debido a la falta de pruebas de diagnóstico, por lo que son necesarios estudios retrospectivos para entender la dinámica inicial del virus.
¿Qué añade el estudio realizado a la literatura?El virus entró en Santiago Región Metropolitana y en Chile alrededor de un mes antes de lo registrado, el pico de la primera ola se subestimó y se alcanzó 3 semanas antes de lo que se creía.
¿Cuáles son las implicaciones de los resultados obtenidos?Las medidas de contención deben aplicarse cuanto antes, ya que hay muchos casos sin detectar, y deben mantenerse aunque no se observen resultados durante unas semanas.
Miguel Ángel Negrín Hernández.
Declaración de transparenciaEl autor principal (garante responsable del manuscrito) afirma que este manuscrito es un reporte honesto, preciso y transparente del estudio que se remite a Gaceta Sanitaria, que no se han omitido aspectos importantes del estudio, y que las discrepancias del estudio según lo previsto (y, si son relevantes, registradas) se han explicado.
Contribuciones de autoríaD. García-García y M.I. Vigo concibieron y diseñaron el estudio. J. Márquez recogió y procesó los datos, y escribió el primer borrador. J. Márquez, D. García-García, M.I. Vigo y C. Bordehore participaron en el análisis, la interpretación y la discusión de los resultados. Todas las personas firmantes participaron en la escritura, la revisión y la aprobación de la versión final del artículo.
AgradecimientosLos autores agradecemos al Instituto Sistemas Complejos de Ingeniería (ISCI), Medicina UCH y el Servicio de Salud Metropolitano Central por los datos del estudio de seroprevalencia; y al DEIS y al Ministerio de Ciencias por los datos de infectados y muertes.
FinanciaciónJ. Márquez ha recibido una beca de doctorado otorgada por la Dirección de Cooperación Internacional de la Universidad Viña del Mar, Chile.
Este estudio ha estado parcialmente financiado por la Universidad de Alicante (contrato COVID-19 2020-41.30.6P.0016) y parcialmente realizado en el Laboratorio Marino UA-Dénia https://web.ua.es/es/marlabdenia/ (Acuerdo Ajuntament de Dénia-Conselleria de Medio Ambiente, Agua, Infraestructuras y Territorio de la Generalitat Valenciana).
Conflictos de interesesNinguno.